
时间:2024-08-22 19:56:33 阅读:610

诺维医学科研官网:https://www.newboat.top
bilibili:文章对应的讲解视频在此。熊大学习社 https://space.bilibili.com/475774512
Gitee开源:https://gitee.com/shenghuaxiong/ioter
CSDN玩转物联网专栏文章:https://blog.csdn.net/shx13141/category_11669532.html
微信公众号:熊大学习社、诺维之舟
公益网站,https://med.newboat.top ,内有医学资料库
课程相关资料:
(1)学习资料,包括CHARLS离线数据库、SCI论文思路复现代码-基于R。关注公众号“熊大学习社”,回复“charls01”,获取资料链接。
谢谢您的支持,我们坚持学以致用、高效学习、质量服务,做好有质量的分享。
另外,服务合作请联系: 见客服二维码。
关注B站熊大学习社,公众号诺维之舟、熊大学习社。您的一键三连是我最大的动力。
1 CHARLS离线数据库,资源获取。
2 CHARLS数据介绍,有哪些年份和分类,有哪些表、哪些数据,怎么阅读和查找。
3 数据提取,基于R,代码实现。
4 数据合并和清洗,基于R,代码实现,最后做一个基线分析。
这次直播课程的特点:手把手撸代码,随时互动交流,拉近和大家的距离,建立研究亲切感。
官网地址:2020年全国追踪调查
中国健康与养老追踪调查(China Health and Retirement Longitudinal Survey,CHARLS) 是由北京大学国家发展研究院主持展开,是国家自然科学基金委资助的重大项目。是一项持续的纵向调查,冒在收集一套代表中国45岁及以上中老年人家庭和个人的高质量微观数据,社会、经济和健康状况,用以分析我国人口老龄化问题,推动老龄化问题的跨学科研究。
https://charls.charlsdata.com/pages/Data/2020-charls-wave5/zh-cn.html
可自行注册账号,申请下载,需要审核通过后才能下载!

CHARLS离线数据库|干净原版|2020年最新,已放学习资料包。也可自行获取:https://nwzz.xyz/buy/28
一共三个版本:
(1)Excel
(2)SPSS
(3)Stata
特点:案例均来源于中国,更适合中国的科研工作者;无需实验验证;最新一期包含疫情相关模块数据;数据公开。
不足:统计时间相对较短,2011年开始;第五轮数据受新冠疫情影响;肿瘤疾病数据少。
使用权限:需工作单位或学校邮箱申请;在致谢中添加相关内容。
基线调查于2011-2012年在全国28个省、150个地区、450个村庄/城市社区开展,每两年追踪一次,目前已有5期数据, 最新一期为2020年数据(2023年11月16日公布)。
CHARLS是中国首个具有全国代表性的中老年人口调查它以美国健康与退休研究(HRS)为蓝本,吸收和参考全球相关老龄调查的设计,比如英国、欧洲、日本、印度和韩国等。
CHARLS的访问应答率和数据质量在世界同类项目中位居前列。
CHARLS各轮调查均获得了北京大学生物医学伦理委员会的批准。
CHARLSI问卷内容:个人基本信息,家庭结构和经济支持,健康状况,体格测量,医疗服务利用和医疗保险,工作、退休和养老金、收入、消费、资产,以及社区基本情况等。
(1)数据表有哪些,以2020年数据为例。

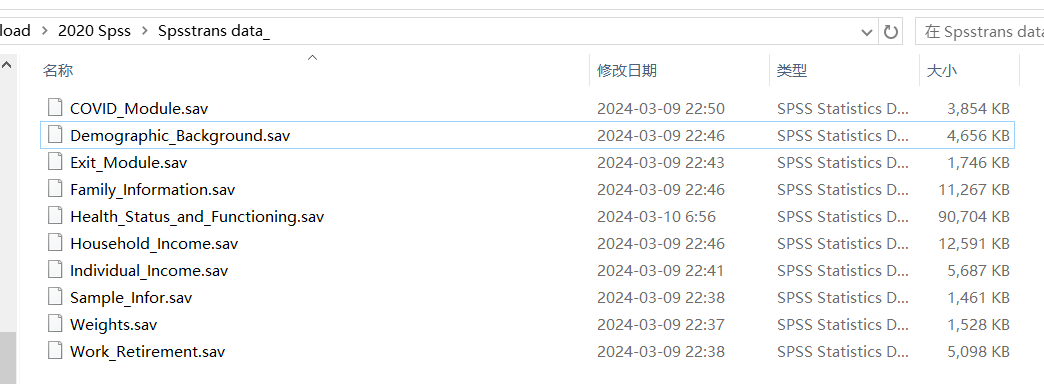

(2)数据项有哪些
查阅CHARLS_2020_Codebook,了解数据所表示的含义。文件在学习资料包。

共42列数据。出生年月和出生地;居住和移民;户口信息;当前工作状态;当前工作的详细信息;失业和求职 教育;婚烟状况及历史。
(1)ba001 ,2020年变量。性别
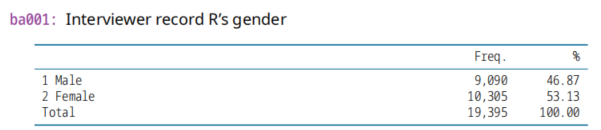
rgender,2011年变量。性别

(2)ba010,2020年变量,教育Education


bd001,2011年变量,教育Education

(3)ba003_1,2020年变量,出生年份,可计算年龄Age
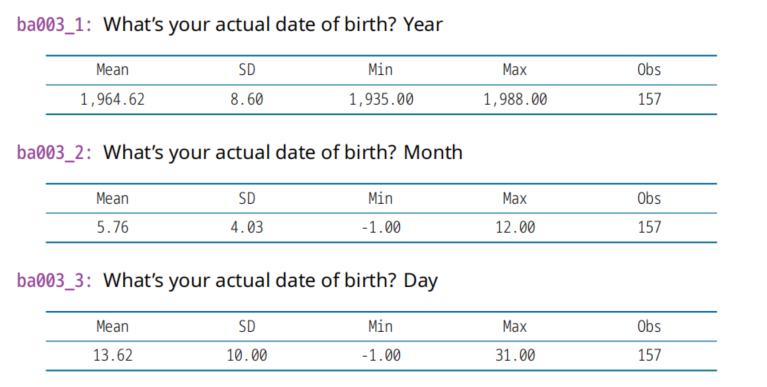
ba002_1 ,2011年变量
从出生年月日到研究当天,计算年龄!
# 计算年龄
# month==0, 按1算
d_demo$ba002_2[d_demo$ba002_2==0] <- 1
# day==0, 按1算
d_demo$ba002_3[d_demo$ba002_3==0] <- 1
# month==2, day==30或29时,将day==28
d_demo$ba002_3[d_demo$ba002_2==2 & (d_demo$ba002_3==30 | d_demo$ba002_3==29)] <- 28
# 出生日期
d_demo$birth <- as.Date(paste(d_demo$ba002_1,d_demo$ba002_2,d_demo$ba002_3, sep='-'))
table(d_demo$ba002_1, useNA = 'ifan') # 54
table(d_demo$ba002_2, useNA = 'ifan')
table(d_demo$ba002_3, useNA = 'ifan')
d1 <- subset(d_demo, is.na(d_demo$birth))
nrow(d1) # 54
# 年龄
d_demo$Age <- round(difftime('2021-3-12', d_demo$birth)/365)
d_demo$Age <- as.numeric(gsub("D", "", d_demo$Age))
(4)ba011,婚姻状况Marriage 2020年变量
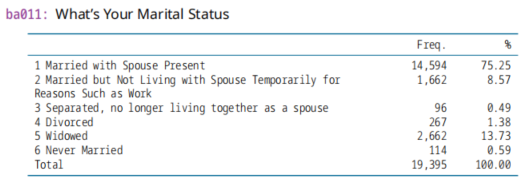
be001,婚姻状况Marriage 2011年变量

(5)place ,户口
ba009:Type of HuKou 2020年变量
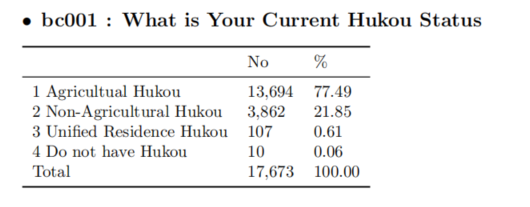
bc001 : What is Your Current Hukou Status 2011年变量
共936列数据。总体健康状况:传染病;生活方式(眼睛,听力,口腔健康,疼痛,骨折);生活行为(睡眠,身体活动,社会联系,饮食,吸烟和饮酒);功能限制;认知(包括数列测试);抑郁症
(1)da046,2020年变量,是否吸过烟,Ever Smoked
(2)da047,2020年变量,现在是否吸烟,Still Smoke or already Quit

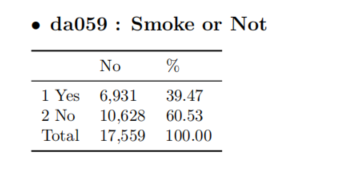
da059: Smoke or Not, 2011年变量
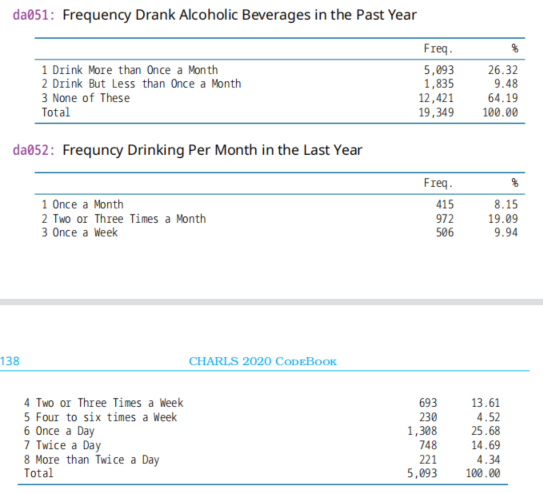
(3)da051,2020年变量,去年饮酒频率,Frequency Drank Alcoholic Beverages in the Past Year
(4)da052,2020年变量,去年每月饮酒频率,Frequncy Drinking Per Month in the Last Year
da067,2011年变量
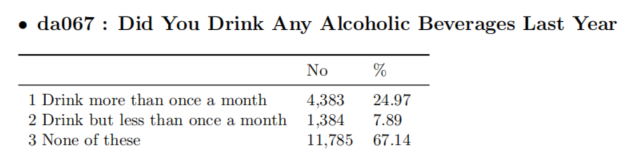
(5)失眠,My Sleep Was Restless
db022,2020年变量
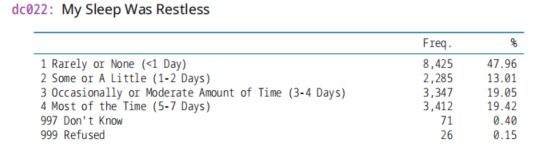
dc015,2011年变量

(6)dc018,2020变量, Felt Depressed,抑郁症
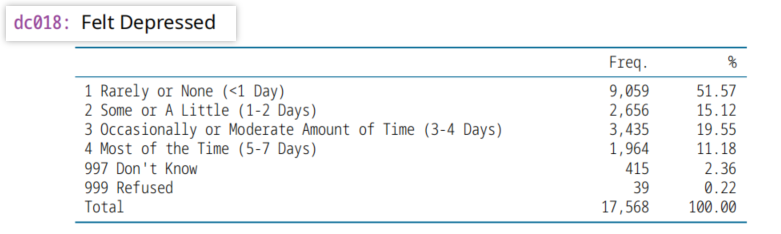
dc011,2011变量

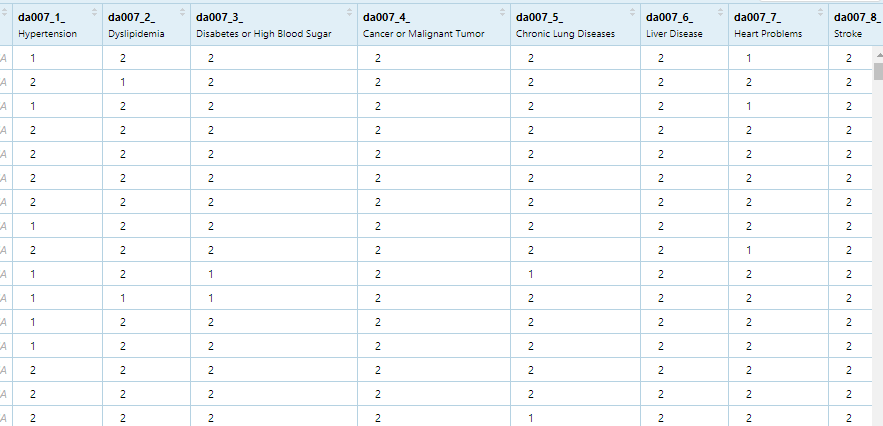
(7)慢性病
其中就包括CVD。CVD全称是Cardiovascular Disease,中文称为心血管疾病。这是一种涵盖心脏和血管系统多种疾病的总称,包括冠心病、高血压、心肌梗塞、心力衰竭等。心血管疾病是全球死因的首位,尤其是在已开发国家和地区。
da007, 2011年变量,
da007_1_ Hypertension 高血压
如下图,依次类推
da007_8_ Stroke 脑卒中,中风

看一下数据
XChroDisType,2020年变量, 慢性病类型
xchrodistype_8_: Name of Disease [8] 中风,脑卒中 Stroke

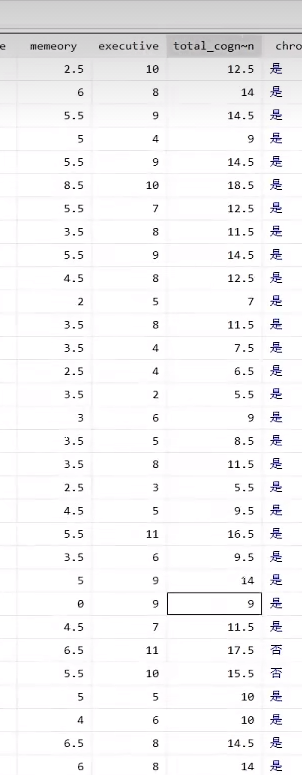
(8)轻度认知障碍cognition
由memeory(情景记忆)和executive(心智状况)综合考虑,按照相应的标准进行计算。
已清洗好的数据,质量好,有需要可联系客户。

说明文档: https://charls.charlsdata.com/pages/Data/2011-charls-wave1/zh-cn.html
(1)qi002,身高

(2)ql002,体重

有了这2个指标,就可以计算BMI
# 表3,biomarkers, 体检
# qi002,身高
# ql002, 体重
d_biom <- read_dta("2011/biomarkers.dta")[,c('ID','qi002','ql002')]
# BMI: bmi = weight_kg / (height_m ** 2)
d_biom$BMI <- round(d_biom$ql002/(d_biom$qi002/100)**2,2)
table(d_biom$qi002, useNA = 'ifan') # 265
table(d_biom$ql002, useNA = 'ifan') # 246
table(d_biom$BMI, useNA = 'ifan')
d1 <- subset(d_biom, is.na(d_biom$BMI))
nrow(d1) # 334
# 删除不需要的列
d_biom <- subset(d_biom, select = -c(qi002,ql002))
(1)newglu, FPG , Glucose, 空腹血糖
(2)newcho, Total Cholesterol , 总胆固醇
(3)newhdl, HDL 高密度脂蛋白 "好的胆固醇"
(4)newldl, LDL 低密度脂蛋白 "坏的胆固醇"
(5)newtg, Triglycerides 甘油三酯
甘油三酯-葡萄糖指数(TyG index)被广泛用作评价胰岛素抵抗的指标。TyG index的计算公式如下:
TyG index = ln [甘油三酯值 (mg/dL) * 空腹血糖值 (mg/dL) / 2]
其中,"ln" 表示自然对数。这个指标的值越高,表示个体的胰岛素抵抗越严重。
# newglu, FPG , Glucose, 空腹血糖
# newcho, Total Cholesterol , 总胆固醇
# newhdl, HDL 高密度脂蛋白 "好的胆固醇"
# newldl, LDL 低密度脂蛋白 "坏的胆固醇"
# newtg, Triglycerides 甘油三酯
d_blood <- read_dta("2011/Blood_20140429.dta")[,c('ID','newglu','newcho','newhdl', 'newldl', 'newtg')]
# TyG指数计算
d_blood$TyG <- round(log(d_blood$newtg * d_blood$newglu/2),2)
table(d_blood$newtg, useNA = 'ifan') # 191
table(d_blood$newglu, useNA = 'ifan')
table(d_blood$TyG, useNA = 'ifan')
d1 <- subset(d_blood[, c('newtg','newglu','TyG')], is.na(d_blood$TyG))
nrow(d1) # 211
共789列数据。预备了17个子女的数据信息,包括子女性别,年龄,婚烟;与主要被访者关系;户口;教育。
ca006_1_,子女性别
ca008_1_, 子女工作状态,工作还是学生
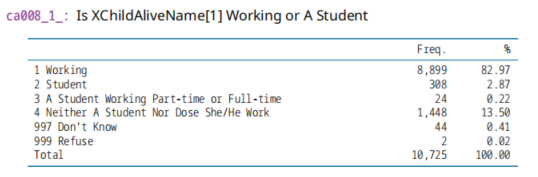
共855列数据。家庭收入和支出;家庭资产;
共210列数据。个人收入及资产
共8列数据。采访时间、是否死亡、是否横断面样本等
共8列数据。家庭权重,个体权重
共160列数据。当前工作状态;当前工作的详细信息;失业和求职活动;退休;养老金。
(1)固体燃料使用,
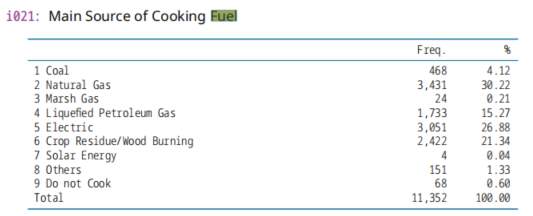
共1129列数据。死亡数据、疾病。
xezdisease_1_,Cancer,癌症
xezdisease_2_,Chronic Lung Diseases,支气管肺病
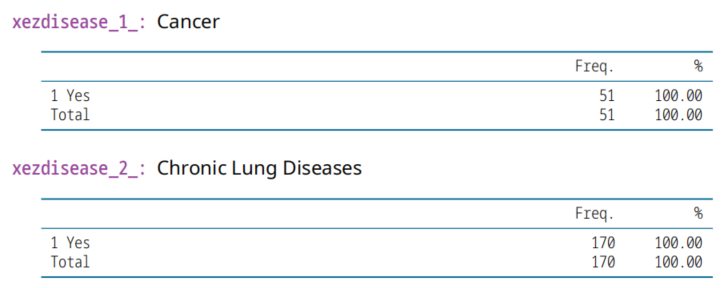
xezdisease_3_: Heart Diseases,心脏病
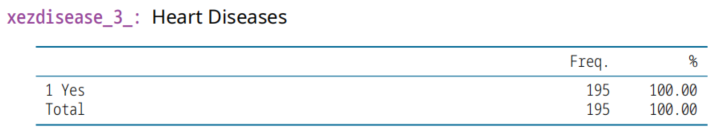
xezdisease_4_,中风, Stroke

exda007,Stroke,中风,脑卒中
exda008,Stroke最近一次时间

共100列数据。疾病防范意识、个人患病和隔离、疫情期间个人活动、疫情期间居住地管控。
va001_s4, Masking, 戴口罩

手把手课程教学,逐行代码实现。
参考文章:The association between triglyceride-glucose index and its combination with obesity indicators and cardiovascular disease: NHANES 2003-2018

我们对应研究:甘油三酯-葡萄糖指数triglyceride-glucose index与脑卒中Stroke的关联性,基于CHARLS
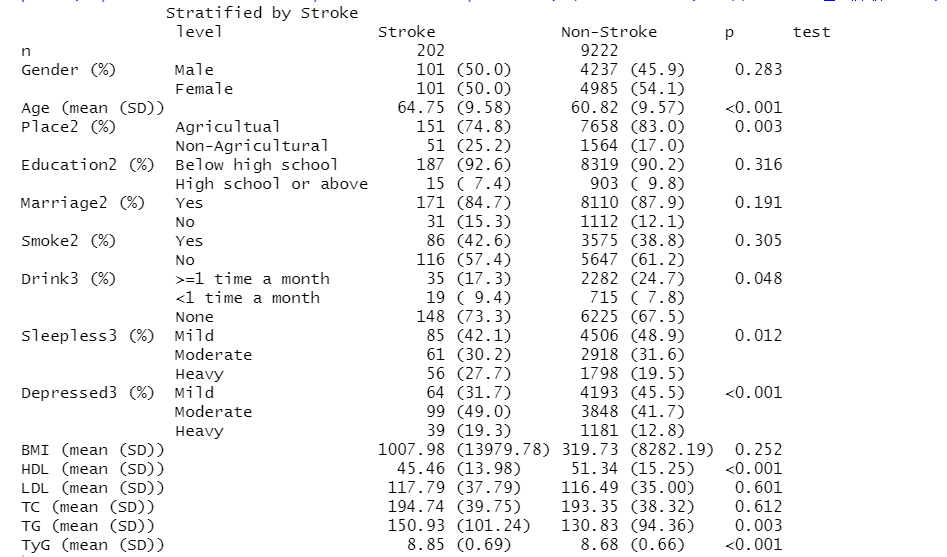
表1,Demographic_Background,人口学
# 表1,demographic_background 人口学
# 第一种方式:导入,Environment->Import Dataset
# 第二种方式:stata, spss的数据均可导入
# demographic_background <- read_dta("2011/demographic_background.dta")
# View(demographic_background)
# 第三种方式:
d_demo <- read_dta("2011/demographic_background.dta")[,c('ID','rgender','ba002_1','ba002_2','ba002_3','bc001','bd001','be001')]
# ID, indiviudal ID
# householdID,
# communityID
# 性别,rgender
# 年龄,出生年月日,ba002_1 ba002_2 ba002_3
# 户口,Place, bc001
# 教育,bd001
# 婚姻,be001
# 计算年龄
# month==0, 按1算
d_demo$ba002_2[d_demo$ba002_2==0] <- 1
# day==0, 按1算
d_demo$ba002_3[d_demo$ba002_3==0] <- 1
# month==2, day==30或29时,将day==28
d_demo$ba002_3[d_demo$ba002_2==2 & (d_demo$ba002_3==30 | d_demo$ba002_3==29)] <- 28
# 出生日期
d_demo$birth <- as.Date(paste(d_demo$ba002_1,d_demo$ba002_2,d_demo$ba002_3, sep='-'))
table(d_demo$ba002_1, useNA = 'ifan') # 54
table(d_demo$ba002_2, useNA = 'ifan')
table(d_demo$ba002_3, useNA = 'ifan')
d1 <- subset(d_demo, is.na(d_demo$birth))
nrow(d1) # 54
# 年龄
d_demo$Age <- round(difftime('2013-3-12', d_demo$birth)/365)
d_demo$Age <- as.numeric(gsub("D", "", d_demo$Age))
# 删除不需要的列
d_demo <- subset(d_demo, select = -c(ba002_1,ba002_2,ba002_3,birth))
# 列名变更
d_demo <- plyr::rename(d_demo, c(rgender='Gender', bc001='Place', bd001='Education', be001='Marriage'))
table(d_demo$Place, useNA = 'ifan')
表2,Health Status and Functioning 健康状况与功能
# 表2,health_status_and_functioning 健康状况
# ID
# 吸烟,Smoke, da059
# 喝酒,Drink, da067
# 失眠,Sleepless, dc015
# 抑郁,Depressed, dc011
# 脑卒中, Stroke, da007_8_
d_health <- read_dta("2011/health_status_and_functioning.dta")[,c('ID','da059','da067','dc015','dc011','da007_8_')]
table(d_health$da059, useNA = 'ifan')
table(d_health$da067, useNA = 'ifan')
table(d_health$dc015, useNA = 'ifan')
table(d_health$dc011, useNA = 'ifan')
table(d_health$da007_8_, useNA = 'ifan')
# 列名变更
d_health <- plyr::rename(d_health, c(da059='Smoke', da067='Drink', dc015='Sleepless', dc011='Depressed', da007_8_='Stroke'))
表3,biomarkers, 体检
# 表3,biomarkers 体检数据
# ID
# 身高,qi002
# 体重,ql002
d_biom <- read_dta("2011/biomarkers.dta")[,c('ID','qi002','ql002')]
# BMI: bmi = weight_kg / (height_m ** 2)
d_biom$BMI <- round(d_biom$ql002/(d_biom$qi002/100)**2,2)
table(d_biom$ql002, useNA = 'ifan')
table(d_biom$qi002, useNA = 'ifan')
d1 <- subset(d_biom, is.na(d_biom$BMI))
# 删除不需要的列
d_biom <- subset(d_biom, select = -c(ql002,qi002))
表4,Blood, 血检
# 表4,Blood_20140429 血检数据
# ID
# HDL, newhdl
# LDL, newldl
# TC, newcho, Total Cholesterol , 总胆固醇
# TG, newtg ,甘油三酯
# FPG,newglu , Glucose, 空腹血糖
d_blood <- read_dta("2011/Blood_20140429.dta")[,c('ID','newhdl','newldl','newcho','newtg','newglu')]
# TyG
d_blood$TyG <- round(log(d_blood$newtg * d_blood$newglu/2),2)
table(d_blood$newtg, useNA = 'ifan')
table(d_blood$newglu, useNA = 'ifan')
table(d_blood$TyG, useNA = 'ifan')
手把手课程教学,逐行代码实现。
(1)数据合并
##1.2 数据合并------
d <- left_join(d_demo, d_health, by=c('ID'))
d <- left_join(d, d_biom, by=c('ID'))
d <- left_join(d, d_blood, by=c('ID'))
# 保存数据
write.csv(d, '..d_raw.csv')
# 下次直接从这里开始
d <- read.csv('..d_raw.csv')[-1]
(2)数据纳排
##2.1 数据纳排
nrow(d) # 17705
# (1)暴露因素, TyG指数
d <- subset(d, !is.na(d$TyG))
17705-11636 # 6069
# (2)结局变量,Stroke 脑卒中
d <- subset(d, !is.na(d$Stroke))
11636-11557 # 79
d$Stroke[d$Stroke==1] <- 'Stroke'
d$Stroke[d$Stroke==2] <- 'Non-Stroke'
d$Stroke <- factor(d$Stroke, levels = c('Stroke', 'Non-Stroke'))
table(d$Stroke, useNA = 'ifan')
# (3)协变量, Gender + Age + Education + Place + Marriage + Smoke + Drink + Sleepless + Depressed + BMI + HDL + LDL + TC
# 性别
d$Gender[d$Gender==1] <- 'Male'
d$Gender[d$Gender==2] <- 'Female'
d$Gender <- factor(d$Gender, levels = c('Male', 'Female'))
table(d$Gender, useNA = 'ifan')
# 教育,分为Below high school和High school or above
d$Education2[d$Education<=5] <- 'Below high school'
d$Education2[d$Education> 5] <- 'High school or above'
d$Education2 <- factor(d$Education2, levels = c('Below high school', 'High school or above'))
table(d$Education2, useNA = 'ifan')
# 婚姻,分为在婚与未婚
d$Marriage2[d$Marriage<=2] <- 'Yes'
d$Marriage2[d$Marriage> 2] <- 'No'
d$Marriage2 <- factor(d$Marriage2, levels = c('Yes', 'No'))
table(d$Marriage2, useNA = 'ifan')
# 户口,分为 Agricultual, Non-Agricultural
d$Place2[d$Place==1] <- 'Agricultual'
d$Place2[d$Place==2] <- 'Non-Agricultural'
d$Place2 <- factor(d$Place2, levels = c('Agricultual', 'Non-Agricultural'))
table(d$Place2, useNA = 'ifan')
# 吸烟,分为Yes/No
d$Smoke2[d$Smoke==1] <- 'Yes'
d$Smoke2[d$Smoke==2] <- 'No'
d$Smoke2 <- factor(d$Smoke2, levels = c('Yes', 'No'))
table(d$Smoke2, useNA = 'ifan')
# 喝酒,分为>=1 time a month, <1 time a month, None
d$Drink3[d$Drink==1] <- '>=1 time a month'
d$Drink3[d$Drink==2] <- '<1 time a month'
d$Drink3[d$Drink==3] <- 'None'
d$Drink3 <- factor(d$Drink3, levels = c('>=1 time a month', '<1 time a month', 'None'))
table(d$Drink3, useNA = 'ifan')
# 失眠,Mild , Moderate, Heavy
d$Sleepless3[d$Sleepless==1] <- 'Mild'
d$Sleepless3[d$Sleepless==2] <- 'Moderate'
d$Sleepless3[d$Sleepless==3] <- 'Moderate'
d$Sleepless3[d$Sleepless==4] <- 'Heavy'
d$Sleepless3 <- factor(d$Sleepless3, levels = c('Mild', 'Moderate', 'Heavy'))
table(d$Sleepless3, useNA = 'ifan')
# 抑郁,Mild , Moderate, Heavy
d$Depressed3[d$Depressed==1] <- 'Mild'
d$Depressed3[d$Depressed==2] <- 'Moderate'
d$Depressed3[d$Depressed==3] <- 'Moderate'
d$Depressed3[d$Depressed==4] <- 'Heavy'
d$Depressed3 <- factor(d$Depressed3, levels = c('Mild', 'Moderate', 'Heavy'))
table(d$Depressed3, useNA = 'ifan')
d <- subset(d, !is.na(d$Gender) & !is.na(d$Age) & !is.na(d$Education2) & !is.na(d$Place) & !is.na(d$Marriage2) &
!is.na(d$Smoke2) & !is.na(d$Drink3) & !is.na(d$Sleepless3) & !is.na(d$Depressed3) & !is.na(d$BMI) &
!is.na(d$Gender) & !is.na(d$Gender) & !is.na(d$Gender) & !is.na(d$Gender) & !is.na(d$Gender) &
!is.na(d$HDL) & !is.na(d$LDL) & !is.na(d$TC) & !is.na(d$Place2))
11557-9424 # 2133
(3)基线分析
加载库
library(tableone)
基线分析,结局变量
##2.2 基线分析
str(d)
var <- c('Gender', 'Age', 'Place2', 'Education2', 'Marriage2', 'Smoke2', 'Drink3', 'Sleepless3', 'Depressed3',
'BMI', 'HDL', 'LDL','TC','TG', 'TyG')
t1 <- CreateTableOne(vars = var, strata = c('Stroke'), data =d)
print(t1, showAllLevels = TRUE, nonnormal = '', smd = F) |> write.csv('..Table 1 基线分析.csv')
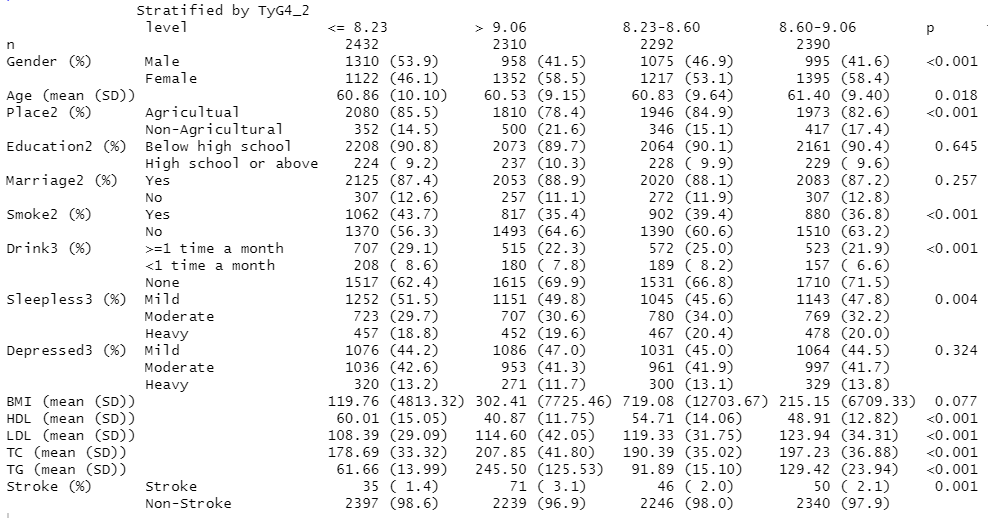
基线分析,暴露变量
# 暴露变量
d$TyG4 <- cut(d$TyG, breaks = 4, labels = c("Q1", "Q2", "Q3", "Q4"))
quantile(d$TyG)
d$TyG4_2[d$TyG <= 8.23] <- '<= 8.23'
d$TyG4_2[d$TyG > 8.23 & d$TyG <= 8.60] <- '8.23-8.60'
d$TyG4_2[d$TyG > 8.60 & d$TyG <= 9.06] <- '8.60-9.06'
d$TyG4_2[d$TyG > 9.06] <- '> 9.06'
table(d$TyG4_2)
var <- c('Gender', 'Age', 'Place2', 'Education2', 'Marriage2', 'Smoke2', 'Drink3', 'Sleepless3', 'Depressed3',
'BMI', 'HDL', 'LDL','TC','TG','Stroke')
# 结局变量
t2 <- CreateTableOne(vars = var, strata = c('TyG4_2'), data =d)
print(t2, showAllLevels = TRUE, nonnormal = '', smd = F) |> write.csv('..Table 1 基线分析2.csv')
(1)公开课抛砖引玉,还有很多变量需要研究。
认知功能账号
环境颗粒物
尿酸
肌肉减少症
(2)课程福利,助力学员零基础成为CHARLS专家,SCI期刊、毕业论文不在话下。

(3)课程资料获取。学习资料,包括CHARLS离线数据库、SCI论文思路复现代码-基于R。关注公众号“熊大学习社”,回复“charls01”,获取资料链接。
谢谢您的支持,我们坚持学以致用、高效学习、质量服务,做好有质量的分享。
另外,服务合作请联系: 见客服二维码。
关注B站熊大学习社,公众号诺维之舟、熊大学习社。您的一键三连是我最大的动力。
CHARLS数据提取与清洗(直播课)学习资料,包括CHARLS离线数据库、SCI论文思路复现代码-基于R、参考文献、讲义。关注公众号“熊大学习社”,回复“charls01”,获取资料链接。